2.2. Результати та їх обговорення
2.2.1. ДНК-аналіз мутацій R408W, IVS12nt1, R158Q, Y414C, IVS10nt546 гена ФАГ
На першому етапі ми вивчали мутації R408W, Y414C (12-й екзон), R158Q (5-й екзон), Ivs10nt546 (10-й інтрон), Ivs12nt1 (12-й інтрон) гена ФАГ у родинах хворих на ФКУ з різних регіонів України. Молекулярно-генетичний аналіз мутацій проводився відповідно до методики описаної в розділі 2.2.
Мутація R408W, локалізована в 12-м екзоні і являє собою одиничну нуклеотидну заміну цитозина на тімін, у результаті чого триплет CGG, що кодує амінокислоту аргінін у 408 положенні замінюється на триплет TGG, що кодує амінокислоту триптофан [74].
Загальний принцип ідентифікації мутації R408W представлений на рис. 2.1. Як видно з малюнків 2.1 і 2.2, при аналізі мутація R408W ми спостерігали три набори рестрикційних фрагментів:
1) два фрагменти розміром 148 пн. і 97 пн. свідчили про наявність сайту рестрикції на обох хромосомах, тобто про присутність мутації в гомозиготному стані;
2) три фрагменти розміром 245 пн., 148 пн. і 97 пн., відповідає сайту рестрикції на одній хромосомі - така рестрикційна картина характерна гетерозиготним носіям мутації R408W;
3) один фрагмент розміром 245 пн. відповідає сайтам рестрикції на обох хромосомах, що адекватно відсутності мутації R408W.
У результаті проведеного дослідження в 11 пробандів з 12 обстежених була виявлена мутація R408W. У шести пробандів мутація виявлена в гомозиготному стані, у п'ятьох у гетерозиготному стані, в одного пробанду мутація R408W виявлена не була. ДНК-аналіз батьків показав наявність мутації в гетерозиготному стані в 17 з 24 обстежених (Рис. 2.2.).
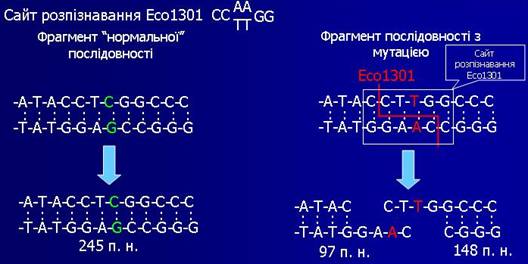
Рис. 2.1. Схема рестрикційного аналізу для детекції мутацій R408W (ендонуклеази рестрикції Eco1301).
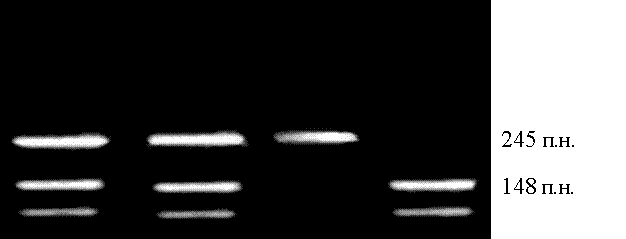
Рис. 2.2. ДНК - аналіз мутації R408W гена ФАГ.
1.8%-ний агарозний гель. 1- мати; 2- батько; 4- пробанд; 3- проба без мутації.
Таким чином, мутація R408W була виявлена на 34 з 48 хромосом 12 з мутаціями гена ФАГ, тобто частота мутації R408W у проаналізованій групі складає 70 % від всіх інших мутацій. Отримані результати свідчать про те, що мутація була виявлена в 11 проаналізованих родинах, тобто в 91.6 % від загальної кількості родин, причому в 50 % мутації були ідентифіковані на обох гомологічних хромосомах 12. Приблизно в 41% випадків мутація була ідентифікована на одній хромосомі з групи хромосом 12 і лише в 9 % випадків мутації, які служили причиною розвитку захворювання, залишилися не ідентифікованими.
Для аналізу мутації R158Q (CGG>СА, аргінін>глутамін), фрагмент 5-го екзона гена ФАГ ампліфікували in vitro з використанням праймера, що вводить одиночну нуклеотидну заміну до складу ампліфікованого фрагмента, у результаті чого створюється сайт рестрикції для ендонуклеази Msp. При мутації R158Q сайт рестрикції зникає (рис. 2.3 і 2.4) [70].
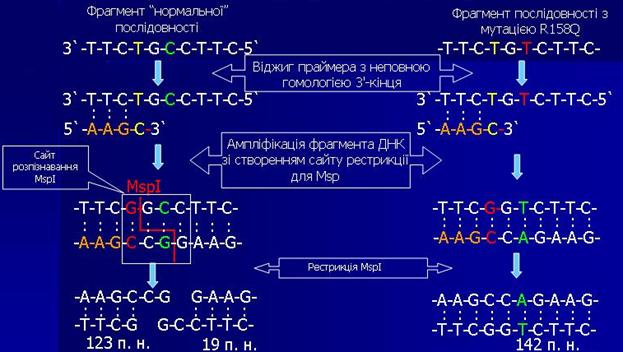
Рис. 2.3. Схема рестрикційного аналізу для детекції мутацій R158Q (ендонуклеази рестрикції Msp).
|
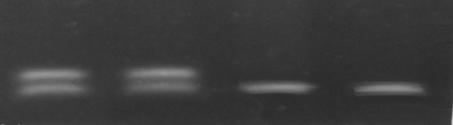
Рис. 2.4. Аналіз мутації R158Q. 1,8 % агарозний гель. 1, 2 - R158Q/норма; 3 - норма/норма, 4 – котроль нома/норма.
Мутація R158Q була ідентифікована нами на 7-ми ФКУ хромосомах. У шести випадках мутація знаходилася в компаунді з R408W і в одному випадку в компаунді з неідентифікованою мутацією. Частота мутації R158Q склала 3,7%.
Мутація Y414C (TAC®TGC), являє собою одиничну нуклеотидну заміну в 12-м екзоні гена ФАГ у результаті, якиї тирозин у 414-м положенні замінюється на цистеін. Як ми бачимо при цій мутації відбувається заміна другого нуклеотиду в складі амінокислотокодуючого триплету. Методика детекції цієї мутації заснована на використанні методу сайт-спрямованого мутагенезу, при якому в процесі ПЛР до складу ампліфікованого фрагмента за допомогою праймера з неповною гомологією на 3'-кінці вводиться одиночна нуклеотидная заміна, що створює сайт рестрикції для ендонуклеази Rsa. При мутації цей сайт зникає, що і є маркером мутації (рис. 2.5) [70].Мутація IVS10nt546, заміна G на А в положенні 546 у 10-м інтроні гена ФАГ, на відстані 11 п.н. від границі 10 інтрон/11 екзон. На відміну від усіх попередніх мутацій, IVS10nt546 локалізована в послідовності інтрона і не спричиняє амінокислотних замін, але приводить до порушення сплайсингу. Нуклеотидна заміна, що виникає при цій мутації, приводить до виникнення сайту для ендонуклеази Dde у послідовності 10-го інтрона, що і служить маркером, що використаний при аналізі мутації (рис. 2.6) [73].
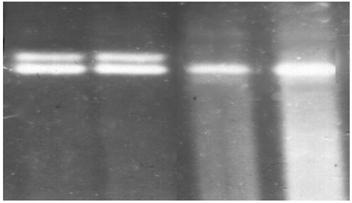
|
Рис. 2.5. Аналіз мутації Y414C. 1,8% агарозний гель. 1, 2 – Y414C/норма; 3 – норма/норма, 4 – контроль, норма/норма.
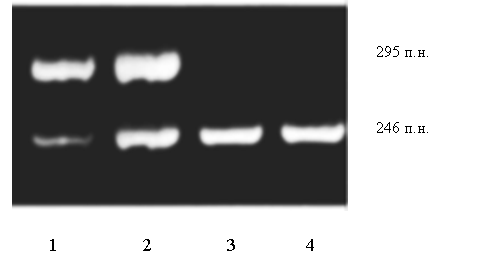
Рис. 2.6. Аналіз мутації Ivs10nt546. 1,8 % агарозний гель. 1, 2 – Ivs10nt546/норма; 3, 4 - норма/норма
У проаналізованій нами групі три пацієнти були носіями даної мутації в компаунді з мутацією R408W.
Мутація IVS12nt1 являє собою одиничну нуклеотидну заміну G на А в послідовності сайту сплайсингу в 12-ом інтроні [75]. Так як і попередня мутація Ivs10nt546, Ivs12nt1 відноситься до групи мутацій сайтів сплайсингу. Маркером цієї мутації є сайт рестрикції ендонуклеази Rsa, що створюється в «нормальній» ампліфікованій послідовності при участі праймера з одиничною нуклеотидною заміною на 3`-кінці і зникає при мутації IVS12nt1 (рис. 2.7) [71].
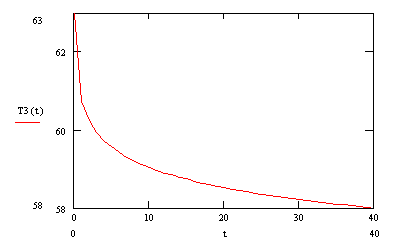
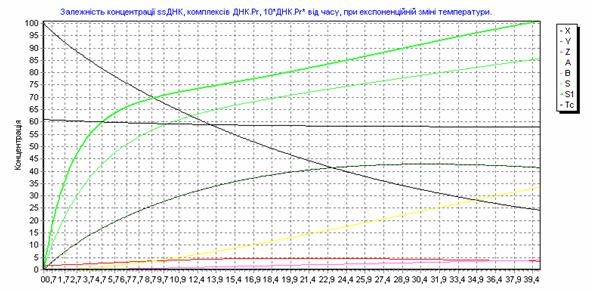
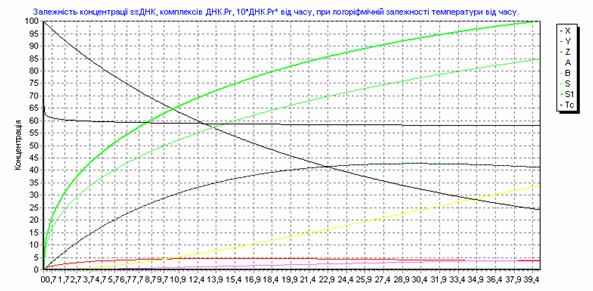
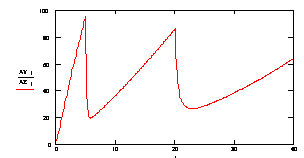
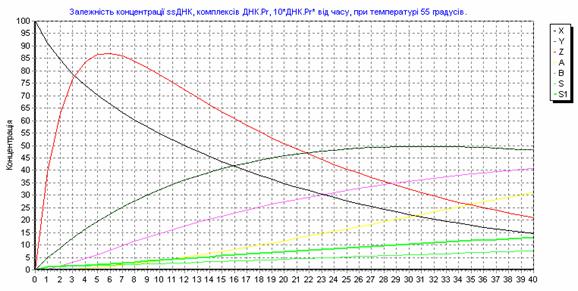
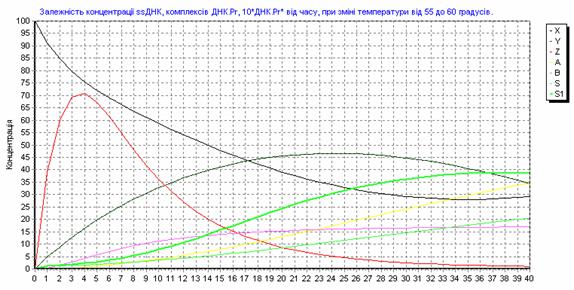
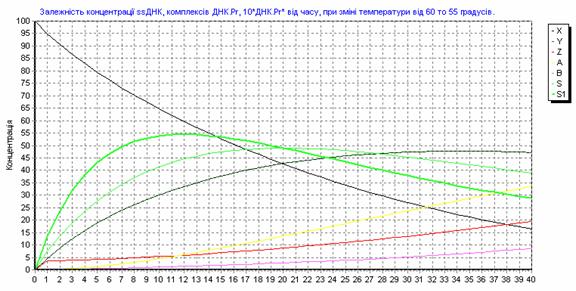
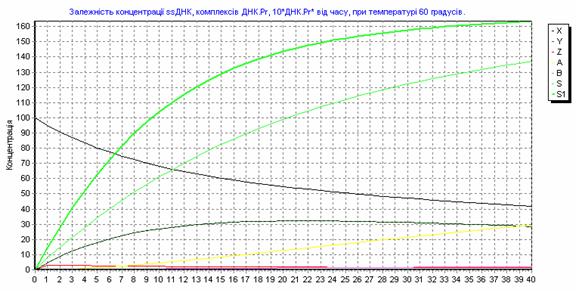
215 п.н. 195 п.н. Рис. 2.7. Аналіз мутації Ivs12nt1.1,8% агарозний гель. 1 - маркер молекулярної маси Ladder 100 bp , 2 – Ivs12nt1/норма; 3, 4 – норма/норма. 2.2.2. ДНК-аналіз мутацій у послідовності 7-го екзона гена ФАГ На сьогоднішній день у 7-му екзоні гена ФАГ ідентифіковано 69 мутацій [37], тому наступний етап нашої роботи був скринінг мутантних варіантів саме у цьому екзоні. На основі аналізу літературних даних нами були підібрані шість найбільш розповсюджених мутацій послідовності сьомого екзона гена ФАГ: P281L, G272X, S273F, R261Q, R261X, R252W, які було доцільно протестувати в пацієнтів [82, 83]. Умови проведення полімеразної ланцюгової реакції представлені в розділі 2.2. У процесі проведення ампліфікації ми зустрілися з проблемою високого виходу неспецифіки, тому були змушені піти по шляху оптимізації умов проведення ампліфікації. У першу чергу була зроблена спроба збільшити температуру ампліфікації з 55ºС до 57 ºС, а потім до 58ºС. При 57ºС неспецифіка, як і раніше, була присутня, а при 58ºС ПЛР продукт був відсутнім. Потім ми використовували багатоступінчастий віджиг праймерів з елементами touch-down ПЛР: 1) попередня денатурація 5 хв при температурі 94ºС; 2) 5 циклів, кожний з яких складається з трьох етапів: - денатурація ДНК – 94 ºС протягом 40 з; - віджиг праймерів - 59 ºС протягом 40 с. - віджиг праймерів - 61 ºС протягом 40 с. - елонгація 72 ºС протягом 50 с., 3) 30 циклів, кожний з яких складається з трьох етапів; - денатурація ДНК – 94 ºС протягом 40 з; - віджиг праймерів - 57 ºС протягом 45 с. - віджиг праймерів - 59 ºС протягом 20 с. - віджиг праймерів - 60 ºС протягом 20 с. - елонгація 72 ºС протягом 50 с., 4) елонгація 72ºС протягом 7 хв. Ампліфікація, що пройшла з цим температурним циклом, мала отримано високу концентрацію специфічного ПЛР продукту при відсутній неспецифічності. Мутація R252W (CGG®TGG), приводить до синтезу білка, у якого аргінін, розташований у 252-м положенні, замінений на триптофан. Для детекції цієї мутації ПЛР продукт 7-го екзона гена ФАГ обробляли ендонуклеазою Eco881, сайт рестрикції для якої зникає при мутації (рис. 2.8) [76]. Рис. 2.8. Аналіз мутації R252W. 1,8% агарозний гель. 1 – R252W/норма; , 2 – норма/норма. 3 - маркер молекулярної маси Ladder 100 bp. Дана мутація була виявлена нами в 3-х пацієнтів: у чотирьох випадках у компаунді з мутацією R408W і в одному випадку в компаунді з неідентифікованою мутацією. Її частота склала 2,9 %. Аргінін у 261-м положенні, утворюючи водневі зв'язки з іншими амінокислотами, виконує функцію стабілізації структури активного центру. Для цього амінокислотного залишку в послідовності білка відомі три мутації R261Q (CGA Мутація R261Q була виявлена в двох пацієнтів. В одного пацієнта в компаунді з мутацією R408W, а в другого в компаунді з неидетифікованною нами мутацією. Таким чином, нами було показано, що дана мутація зустрічається в Україні з частотою 1,2 %. Мутація R261X виявлена не була. Мутація S273F (TCC(TTC) приводить до порушення структури активного центра. При заміні цистеіна на тімін у положенні 273 у послідовності 7-го екзону гена ФАГ формується сайт рестрикції для ендонуклеази Sau3A, що і служить маркером мутації, що був використаний для її детекції [75]. Дана мутація була виявлена в одного пацієнта в компаунді з мутацією R413P. Мутацію S273F поширено в Україні з частотою 0,6%. Амінокислотний залишок пролина в 281-м положенні є останньою амінокислотою послідовності 7-го екзона. Кодується дана амінокислота триплетом CCg, що при мутації P281L замінюється на триплет CTg, що відповідає лейцину. Для ідентифікації даної мутації ПЛР продукт 7-го екзона гена ФАГ, синтезований з використанням методу сайт спрямованого мутагенезу, гідролізують ендонуклеазою Msp, сайт дізнавання для якого формується в процесі ПЛР на «нормальній» послідовності гена ФАГ, але є відсутнім при мутації P281L (рис. 2.9) [77]. Мал. 2.9. Аналіз мутації P281L. 1,8 % агарозний гель. 1 - P281L/норма; 2, 4 - норма/норма, 3 - маркер молекулярної маси Ladder 100 bp. У проаналізованій нами групі дана мутація була виявлена в двох пацієнтів. В обох випадках у компаунді з мутацією R408W. Частота мутації P281L у досліджуваній популяції склала 2,3 %. Мутація G272X – відноситься до класу нонсенсів-мутацій. Розташований у 272-м положенні гуанін замінюється на стіп кодон, що термінирує транскрипцію гена ФАГ і приводить до синтезу транскрипта, в який відсутні останні 6 екзонів. Для детекції мутації G272X ПЛР продукт 7-го екзона гена ФАГ гідролізують ендонуклеазою BamHI, сайт рестрикції для якого відсутній у мутантній послідовності. 2.2.3. Аналіз алельного поліморфізму VNTR-локусу 3`-нетрансльованої області гена ФАГ. Аналіз алельного поліморфізму VNTR-локусу 3'- нетрансльовані області гена ФАГ проводили серед 68 здорових донорів з різних регіонів України. У результаті дослідження було виявлено 7 алельних варіантів мінісателітного VNTR-локусу. Число тандемних повторів при цьому варіювало від 3 до 12, а розміри продуктів ампліфікації варіювали від 380 до 650 п.н. відповідно числу тандемних повторів. На рис. 2.10 представлені деякі алельні варіанти VNTR-локусу. 1 2 3 4 М Рис. 2.10. Електрофореграмма продуктів ампліфікації мінісателлітного VNTR –поліморфізму, 1.8% агарозний гель: 1 - генотип 3/3 (380/380 п.н.); 2 - генотип 3/7 (380/500 п.н.); 3 - генотип 3/8 (380/530 п.н.); 4 - генотип 9/12 (560/650 п.н.); M - Маркер молекулярної маси ДНК плазміди pBR322 гідролізована ендонуклеазою Msp. 2.2.4. Оптимізація умов проведення ампліфікації Об'єктом оптимізації може виступати тест-система для діагностики фенілкетонурії, а також інші тест системи. Основною проблемою при ампліфікації ДНК in vitro є її неспецифіка. Специфічність ампліфікації – це відношення концентрації цільового ПЛР продукту до всього ПЛР продукту. На специфічність ампліфікації визначеної послідовності ДНК впливають наступні фактори: 1) Специфічність відпалу праймерів. 2) Помилка в роботі полімерази. 3) Повнота денатурації ланцюгів ділянки ДНК матриці під час подовження ланцюга. Основним напрямком оптимізації ампліфікації було збільшення специфічності відпалу праймерів при високих значеннях виходу продукту. 2.2.4.1. Специфічність відпалу праймерів. Віджиг праймерів може проходити в різних локусах ДНК матриці. При виборі праймерів вибирають такі праймери, що мають тільки одну унікальну ділянку відпалу на матриці. Як буде показано нижче, праймери можуть обжигатися і на не цілком комплементарній матриці. Віджиг праймерів на не цілком гомологічній матриці представлений на рис. 2.11, де перших сім проб - ПЛР з однією парою праймерів, других сім проб - мультиплексна ПЛР. Різна інтенсивність ПЛР продукту може бути пояснена тим, що при ампліфікації з однією парою праймерів немає конкуренції праймерів за ssДНК, а при мультиплексній ПЛР спостерігається конкуренція між праймерами за ssДНК. Ця конкуренція приводить до того, що при мультиплексній ПЛР переважно подовжуються комплекси праймер-ssДНК, які мають максимальну гомологію і, як наслідок, - максимальну стійкість і концентрацію. Це може бути особливо актуально, коли відпалу праймерів з цільовою ділянкою перешкоджає стеричний фактор або інші фактори. У таких випадках збільшують температуру та час відпалу праймерів. Однак збільшення температури відпалу праймерів приводить до зниження інтенсивності ампліфікації (див. рис 1.7.), що обумовлено зниженням різниці енергії Гібса в реакції утворення ДНК.Пр і, як наслідок, - стійкості комплексу ДНК.Пр. Рис. 2.11. Продукти ПЛР, отримані при однакових умовах ПЛР – циклу на сімох різних матрицях. Перші сім проб ПЛР - з однією парою праймерів, других сім проб - мультиплексна ПЛР. У зв'язку з цим дослідники проводять багато східчастий віджиг праймерів при різній температурі. При цьому вони покладаються як на власний досвід, так і на досвід колег. І так, якщо не вдається провести віджиг праймерів при одній температурі, проводять багатоступінчатий віджиг праймерів. При багатоступінчастому відпалу праймерів, як правило, температуру збільшують (див. таблицю 2.5.). Іноді проводять багатоступінчате зниження температури. Таблиця 2.5. Зміна температури під час ампліфікації. Температура ºС Час c 56 40 57 20 58 20 60 30 Подальшою метою буде розгляд переваг і недоліків багатоступінчатого відпалу праймерів. 2.2.4.2. Постановка задачі оптимізації При постановці ампліфікації в нас є два критерії оцінки її ефективності: вихід продукту і специфічність. Ми зацікавлені в максимізації обох критеріїв. Основним інструментом для зміни цих критеріїв є температура відпалу праймерів. Однак будь-яка зміна температури приводить до збільшення одного з параметрів і зниженню іншого. Можливі наступні варіанти зміни температури в часі, які ми розглянемо далі: - температура, що змінюється східчасто; - температура, задана у вигляді функції (2.85 - 2.88). Для вибору оптимального режиму зміни температури відпалу праймерів необхідно задати початкові обмеження: специфічність одного раунду ампліфікації вище 99%, відсоток відпалених праймерів вище 75%. 2.2.4.3. Отримання математичної моделі ампліфікації. Для оцінки переваг і недоліків багатоступінчастого відпалу праймерів необхідно вивести математичну модель ампліфікації. Для цього розглянемо конкурентні процеси, що відбуваються при відпалу праймерів. Виділимо чотири процеси, що протікають при відпалу праймерів, і проаналізуємо їх вплив на специфічність ампліфікації. 1) Утворення комплексу ДНК.Пр ДНК+Пр Швидкість даного процесу, згідно закону діючих мас, описується рівнянням (2.2). V1= де k1 – константа швидкості утворення комплексу ДНК.Пр. Процес проходить зі значним надлишком Пр, тому C(Пр) можна ввести в константу, тоді рівняння прийме вид (2.3). V1= 2) Руйнування комплексу ДНК.Пр: ДНК.Пр Швидкість даного процесу, згідно закону діючих мас, описується рівнянням (2.5). V2= де k2 – константа швидкості руйнування комплексу ДНК.Пр. 3) Утворення комплексу ДНК*.Пр з неповною гомологією. ДНК+Пр Швидкість даного процесу, згідно закону діючих мас, описується рівнянням (2.7). V3= де k3 – константа швидкості утворення комплексу ДНК*.Пр. Процес проходить зі значним надлишком Пр, тому C(Пр) можна ввести в константу, тоді рівняння прийме вид (2.8) V3= 4) Руйнування комплексу ДНК*.Пр з неповною гомологією. ДНК*.Пр Швидкість даного процесу, згідно закону діючих мас, описується рівнянням (2.10). V4= де k4 – константа швидкості руйнування комплексу ДНК*.Пр. Застосовуючи закон збереження маси, одержимо рівняння (2.11), продиференцирувавши яке одержимо рівняння (2.12). З(ДНК)+ Для простоти запису замінимо x = С(ДНК), y = C(ДНК.Пр), z = C(ДНК*.Пр), a = C(ДНК)0 і одержимо: У даному рівнянні k1, k2, k3, k4 = const. Підставимо рівняння (2.18) у рівняння (2.16) і одержимо: Виразимо z з рівняння (2.15) і підставимо в (2.19): Виразимо x і Підставивши x з рівняння (2.22) і Рівняння (2.26) це лінійне неоднородне диференційне рівняння другого порядку. Його загальне рішення можна представити як суму загального рішення однородного рівняння yоо і часткового рішення неоднородного рівняння yчн. Однородне рівняння має вид: Рішення однородного рівняння другого порядку необхідно шукати у виді Знайдемо першу і другу похідну загального рішення однородного рівняння: Підставивши вираження (2.28), (2.29) у вираження (2.27), одержимо вираження (2.30). З огляду на те, що Рівняння (2.31) є квадратним рівнянням, рішенням якого буде рівняння (2.32). Знайшовши корені рівняння (2.31), ми знайшли корені диференціального рівняння другого порядку з однородною правою частиною. Ліва частина рівняння (2.26) константа, тому часткове рішення неоднородного рівняння yчн варто шукати у вигляді константи. З огляду на те, що перша і друга похідна від константи дорівнюють нулю, при підстановці в рівняння (2.26) yчн =const одержимо рівняння (2.34) Для знаходження х необхідно виразити Підставимо знайдене значення y і Підставимо знайдені значення y і z у вираження (1) та виразимо x: Знаходження С1 і С2. З огляду на те, що З огляду на те, що Підставимо знайдені значення С1 і С2 у рівняння (2.40): Підставимо знайдені значення С1 і С2 у рівняння (2.13) та отримаємо рівняння (2.51): Для вираження z підставимо знайдені значення для С1 і С2 у рівняння (2.41) і одержимо рівняння (2.52). Виразимо k2, k3, k4 через k1. Константа швидкості утворення комплексу ДНК.Пр (k1) є функцією від T, властивостями реагуючих речовин і інших параметрів (і) визначається (досвідченим шляхом) для кожної реакції індивідуально. При кожнім ПЛР циклі концентрація ПЦР-продукта збільшується в 1,8 – 1,95 рази, тому ми можемо задати k1=0,05, при якому 85% ДНК буде перетворюватися в комплекс ДНК.Пр. Константа швидкості руйнування комплексу ДНК.Пр ( де K – константа рівноваги. Константа рівноваги може бути розрахована з рівняння (2.54). де Різниця енергій Гібса до і після реакції може бути виражена згідно рівняння (2.33). де Ентальпія й ентропія можуть бути обчислені за принципом адитивності [51] з якої випливає рівняння 2.57 і 2.58. Виразимо Константа швидкості утворення комплексу ДНК.Пр* (k3) залежить від: - імовірності зіткнення двох молекул (ДНК та Пр); - енергетичного бар'єра; - енергії молекул; - (просторових факторів). Компоненти реакційної суміші, які утворюють специфічні та неспецифічні комплекси ДНК.Пр однакові (ДНК і Пр), тому імовірність зіткнення ДНК та Пр, енергетичний бар'єр, енергія молекул і константа швидкості однакові. Тобто Константа швидкості руйнування комплексу ДНК.Пр* ( Тепер, коли ми виразили x, y, z через Рис. 2.12. Залежність концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу, при 60 ºС. Рис. 2.13. Графік зміни швидкості нагромадження комплексу ДНК-праймер ( З графіків 2.12. і 2.13. видно, що віджиг праймерів при температурі 600С (близької до температури плавлення) має переваги і недоліки. До переваг можна віднести те, що віджиг праймерів йде з високою специфічністю. Концентрація комплексів неспецифічного комплексу ДНК - праймер (ДНК.Пр*) дуже низька С(ДНК.Пр*)t=40c = 0.23 %. До недоліків можна віднести те, що швидкість приросту концентрації комплексів ДНК.Пр швидко падає (див. рис. 2.13). Тому нам необхідно змінювати температуру, підтримуючи оптимальну швидкість утворення комплексів ДНК.Пр на тім рівні, що забезпечить потрібний рівень специфічності. Одержимо математичну модель процесу відпалу праймерів. За умови, що температура є функцією від часу, то k2 і k4 також є функціями від часу. Описувати дану систему буде система диференціальних рівнянь: Дане рівняння проблематично вирішити аналітично, його можна вирішити для конкретно заданої функції, наприклад такої як Після підстановки y(0) = y0 у вираз (2.36) одержимо рівняння (2.62). Після підстановки x(0) = x0 у вираз (2.40) одержимо рівняння (2.62). Підставимо знайдені значення С1 і С2 у рівняння (2.36, 2.40, 2.41) і виразимо x, y та z. Тепер ми можемо побудувати графік 2.14. залежності концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу, при температурі що східчасто змінюється 10 секунд при 600С, 30 секунд при 580С. Для побудови графіка використовуємо програму Mathcard 2001 Прofessional. Для порівняння наведемо графік залежності концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу, при температурі 600С (рис. 2.12) та 580С (рис. 2.15). Якщо порівняти малюнки 2.14 та 2.15, то видно, що віджиг праймерів набагато вигідніше здійснювати при температурі, що підвищується, від 58ºС до 60ºС. При відпалу праймерів при температурі, що підвищується з 58ºС до 60ºС відношення концентрацій ДНК.Пр до ДНК.Пр* складе 230, у той час як при зниженні температури від 600С до 580С відношення концентрацій ДНК.Пр до ДНК.Пр* складе 80. Як видно з графіка рис. 2.16, зниження температури від 600С до 580С приводить до збільшення швидкості нагромадження комплексу ДНК.Пр, але в той же час веде до стрімкого збільшення концентрації неспецифічного комплексу ДНК.Пр*. Необхідно відзначити, що при зменьшенні температури, швидкість нагромадження специфічних і неспецифічних комплексів ДНК-праймер збільшується (див. рис. 2.14.). Рис. 2.14. Залежність концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу, при температурі, що змінюється, 10 сек при 60°С, 30сек при 58°С. Рис. 2.15. Залежність концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу, при 580С. Рис. 2.16. Графік зміни швидкості нагромадження комплексу ДНК-праймер ( Розглянемо процес відпалу праймерів при зростаючій температурі. На рис. 2.17. представлений графік залежності концентрації комплексів ДНК.Пр до ДНК.Пр*, від часу при температурі, що збільшується: 10 секунд при температурі 58ºС, 30 секунд при температурі 60ºС. Як видно з рис. 2.17. підвищення температури з 58°С до 60°С призводить до різкого зниження концентрації неспецифічних комплексів ДНК-праймер, що, безумовно, має позитивний ефект, але цей позитивний ефект невелюється різким зниженням швидкості приросту специфічних комплексів ДНК-праймер Рис. 2.18. Рис. 2.17. Залежність концентрації комплексів ДНК.Пр до ДНК.Пр*, від часу при температурі, що змінюється: 10 сек при 58°С, 30 сек при 60ºС. Рис. 2.18. Залежність відносини концентрації комплексів ДНК.Пр до ДНК.Пр*, від часу, при температурі, що змінюється: 10 сек при 58°С, 30 сек при 60°С. Рис. 2.19. Залежність зміни концентрації ssDNA (синя лінія), специфічного комплексу ДНК.Пр (зелена лінія), неспецифічного комплексу ДНК.Пр* (червона лінія) від часу, при температурі, що змінюється, 5 скунд при 60,5ºС, 15 скунд при 58ºС и 20 скунд при 56ºС. Рис. 2.20. Швидкість зміни концентрації комплексу ДНК-праймер у перебігу часу. Рис. 2.21. Зміна відношення концентрації специфічного комплексу ДНК-праймер до неспецифічного комплексу ДНК-праймер у перебігу часу. Перед нами була поставлена ціль збільшення виходу продукту і специфічності. Для максимізації виходу продукту необхідно поступово знижувати температуру відпалу праймерів і тим самим підтримувати швидкість нагромадження специфічного продукту, але знижувати її такими темпами, щоб не допускати зниження специфічності нижче 99%. Розглянемо процес відпалу праймерів при ступінчато зростаючій температурі. На рис. 2.19. представлений графік залежності концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу при температурі, що змінюється: 5 секунд при температурі 60,5ºС, 15 секунд при 58ºС и 20 секунд при 56ºС. Проведення багатоступінчастого відпалу праймерів з поступовим зниженням температури дозволяє підтримувати високу швидкість утворення специфічних комплексів ДНК-праймерів (Рис. 2.20), при високій специфічності (Рис. 2.21). Приведена модель моделює процес відпалу праймерів і розглядає 4 процеси: 1) ssДНК+Пр 2) ssДНК.Пр 3) ssДНК+Пр 4) ssДНК*.Пр Але модель не розглядає процес елонгації ланцюга та ренатурациї ДНК: 5) ssДНК.Пр + dNTP 6) ssДНК*.Пр + dNTP 7) ssДНК + ssДНК Остання реакція (2.78) йде набагато повільніше інших реакцій, тому що C(ssДНК) << C(Пр), і ми не будемо її розглядати. Швидкість утворення ПЛР – продукту лімітується концентрацією ssДНК.Пр та ферменту. Швидкість утворення продукту полімеразної ланцюгової реакції описується рівнянням (2.79). V5=k5.C(ssДНК.Пр) . C(E) (2.79) де: C(E) – концентрація ферменту. С(E) не змінюється в часі, тому ми можемо внести її в k5 і одержати рівняння (2.80) V5=k5.C(ssДНК.Пр) (2.80) Залежність швидкості синтезу від температури представлена в таблиці 1.11. Виходячи з даних таблиці 1.11., ми вивели кореляційне рівняння (2.81), що зв'язує швидкість елонгації ланцюга з температурою, яке буде використано для інтерполяції. k5= 0.02 Виходячи з приведеного вище, побудуємо математичну модель даного процесу. Даний процес буде описуватися системою диференційних рівнянь (2.82). Щоб спростити запис зробимо заміну: x = С(ДНК), y = C(ДНК.Пр), z = C(ДНК*.Пр), A = C(ДНК), B=C(ДНК*), a = C(ДНК)0 і одержимо: У даному рівнянні k2, k4, k5, k6 - функції від температури, а температура є функцією від часу. Аналітичне рішення даного рівняння є дуже проблематичним, тому застосуємо ітераційний метод. Суть даного методу полягає в тому, що ми спочатку задаємо початкові параметри x=100, Після Даний метод має переваги і недоліки. До переваг можна віднести те, що ми можемо задавати температуру як функцію від часу й інших параметрів будь-якого ступеню складності. До недоліків можна віднести меншу точність, чим при аналітичному вирішенні системи диференціальних рівнянь, але це нівелюється за рахунок низького значення dt, що забезпечує високу точність. Проаналізуємо динаміку зміни концентрації компонентів ПЛР системи під час ампліфікації за допомогою програми GraphBuilder при різних функціональних залежностях температури від часу. Результати аналізу наведені в Табл. 2.6. Таблиця 2.6. Концентрації реагентів при різних температурах відпалення праймерів. № Початкова температура Кінцева температура Функція Концентрації реагентів X Y Z A B 1 55 55 14,4 48,2 2,1 31,2 4,1 3 55 60 29.2 34.4 0.1 34.6 1.7 2 60 55 16.4 47.2 2 33,6 0,9 4 60 60 41.6 28.6 0.1 29.5 0.2 5 58 58 21,9 41,9 0,3 35,2 0,65 Якщо врахувати, що при елонгації ланцюга ДНК.Пр і ДНК.Пр* цілком перетворюються у відповідний ПЛР – продукт, то необхідно розглядати не A і B, а A+Y і B+Z. Результати наведені в таблиці 2.7. Таблиця 2.7. Концентрации реагентів при різних температурах відпалення праймерів. № Функція Концентрації реагентів Рис. X A+Y B+Z 1 14,4 79,4 6,18 12,9 2.22 2 29.2 69,0 1,79 38,6 2.23 3 16,4 80,83 2,81 28,74 2.24 4 41.6 58,02 0,35 163,62 2.25 5 21,9 77,13 0,97 79,22 - Рис. 2.22. Залежність концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу при 550С. Рис. 2.23. Залежність концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу, при зміні температури від 55 до 600С. Рис. 2.24. Залежність концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу, при зміні температури від 600С до 550С. Рис. 2.25. Залежність концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу при 600С. При порівняні першого та четвертого рядка таблиці 2.7 видно, що з ростом температури збільшується специфічність і зменшується вихід продукту, що узгоджується з практикою і літературними даними. Наша задача - підібрати такий температурний режим, при якому специфічність була б вище 99%, а вихід продукту максимальний. При цьому доцільно розглядати не тільки лінійну зміну температури (загальне рівняння можна записати у вигляді (2.85)), але й інші функції: - експонентна зміна температури (загальне рівняння можна записати у виді (2.86)); - ступенева зміна температури (2.87); - логарифмічна зміна температури (2.89). При пошуку функції оптимального відпалу праймерів будемо змінювати: - температуру старту відпалу праймерів з температурою від 55°С до 65°С з кроком 1 градус; - кінцеву температуру відпалу праймерів з 55°С до 65°С із кроком 1 градус; - коефіцієнт A, який визначає вид функції зміни температури, для кожної функції міняється по різному. У такий спосіб ми змоделювали процес відпалу праймерів при температурі, що змінюється, згідно функцій (2.86 – 2.89). Інтервали варіювання і крок варіювання температури старту, температури закінчення відпалу праймерів і коефіцієнта А, а також кількість проаналізованих функцій, представлені в таблиці 2.8. Таблиця 2.8. Параметри пошуку функції оптимальності. Вид функції Інтервал варіювання температури старту ºС Крок варіювання температури старту ºС Інтервал варіювання температури закінчення ºС Крок варіювання температури закінчення ºС Інтервал варіювання коефіцієнта А Крок варіювання коефіцієнта А Кількість проаналізова-них функцій (2.86) [55; 65] 1 [55; 65] 1 [-3; 3] 0.1 11.11.60 = 7260 (2.87) [55; 65] 1 [55; 65] 1 [0; 3] 0.1 11.11.30 = 3630 (2.88) [55; 65] 1 [55; 65] 1 (0; 3] 0.1 11.11.30 = 3630 (2.89) [57; 67] 0.2 [57; 67] 0.2 [-2; -0.45] 0.05 50.50.31 =77500 Ми записували кількісні показники стану системи з інтервалов у 1 секунду в базу даних, тому отримали 40 записів на один варіант зміни температури ампліфікації і 3680800 записів на 92020 варіантів зміни температури ампліфікації. Після чого ми виконали скрипт, наданий у додатку 2, який дозволив вибрати ті записи, де специфічність перевищила 99% і вихід продукту перевищив 75%. Результат виконання скрипта наданий у таблиці 2.9. Таблиця 2.9. Концентрации реагентів при різних температурах відпалення праймерів. Параметри функції Концентрація Специфіч-ність Функція Accelera-tion Tsta Tend X ssDNA Y Z A B AY BZ S S1 (2.86) -3,8 61 58 23,7 41,6 0,35 33,8 0,4 75,4 0,75 83,1 100,09 (2.86) -3,7 61 58 23,86 41,6 0,35 33,79 0,4 75,39 0,75 83,59 100,26 (2.86) -3,6 61 58 23,93 41,57 0,35 33,75 0,4 75,32 0,75 84,11 100,43 (2.86) -3,5 61 58 24,01 41,54 0,35 33,7 0,4 75,24 0,75 84,65 100,6 (2.86) -3,4 61 58 24,09 41,51 0,35 33,65 0,4 75,16 0,75 85,19 100,77 (2.86) -3,3 61 58 24,17 41,48 0,35 33,6 0,39 75,08 0,74 85,75 100,92 (2.89) -0,4 59,6 58,2 24,233 40,559 0,30 34,458 0,446 75,01 0,75 77,286 100,00 (2.89) -0,8 61,4 58 24,222 41,162 0,35 33,866 0,397 75,02 0,74 85,266 100,14 (2.89) -0,8 61,6 58 24,243 41,164 0,35 33,845 0,396 75,00 0,74 85,512 100,26 (2.89) -0,75 63 58 24,124 41,211 0,35 33,913 0,4 75,12 0,75 84,689 100,01 (2.89) -0,75 63,2 58 24,127 41,211 0,35 33,911 0,4 75,12 0,75 84,715 100,02 (2.89) -0,75 63,4 58 24,128 41,212 0,35 33,909 0,4 75,12 0,75 84,735 100,03 (2.89) -0,75 63,6 58 24,13 41,212 0,35 33,907 0,4 75,11 0,75 84,75 100,04 (2.89) -0,75 63,8 58 24,131 41,212 0,35 33,906 0,4 75,11 0,75 84,762 100,05 (2.89) -0,75 64 58 24,132 41,212 0,35 33,905 0,4 75,11 0,75 84,771 100,05 (2.89) -0,75 64,2 58 24,132 41,212 0,35 33,905 0,4 75,11 0,75 84,778 100,05 (2.89) -0,75 64,4 58 24,133 41,212 0,35 33,904 0,4 75,11 0,75 84,783 100,06 (2.89) -0,75 64,6 58 24,133 41,213 0,35 33,904 0,4 75,11 0,75 84,787 100,06 (2.89) -0,75 64,8 58 24,133 41,213 0,35 33,903 0,4 75,11 0,75 84,79 100,06 (2.89) -0,75 65 58 24,133 41,213 0,35 33,903 0,4 75,11 0,75 84,792 100,06 З таблиці видно, що найбільший інтерес становлять функції (2.86) і (2.89). У функції (2.86) температура відпалу праймерів знаходиться в експонентній залежності від часу. Графік залежності температури від часу для функції (2.90) представлений на рис. (2.26). Залежність концентрації ssДНК, комплексів ДНК.Пр, ДНК.Пр*, специфічних та не специфічних продуктів ампліфікації, специфічності від часу при зміні температури представлена у формулі (2.90) та наведенна на рис. 2.28. Рис. 2.26. Графік залежності температури від часу для функції (2.90). У функції (2.89) температура відпалу праймерів змінюється в логарифмічній залежності, графік залежності температури від часу для функції (2.91) наведенний на рис. (2.27.). Рис. 2.27. Графік залежності температури від часу для функції (2.91). Залежність концентрації ssДНК, комплексів ДНК.Пр, ДНК.Пр*, специфічних та не специфічних продуктів ампліфікації, специфічності від часу при зміні температури по формулі (2.91) наведенні на рис. 2.29. Рис. 2.28. Залежність концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу при зміні температури по формулі (2.90). Рис. 2.29. Залежність концентрації ssДНК і комплексів ДНК.Пр, ДНК.Пр* від часу при зміні температури по формулі (2.91). Застосування отриманої математичної моделі відпалення праймерів дозволяє проводити відпалення праймерів при високих значеннях специфічності. 3. Висновки. 1. Проведено детекцію мутацій R408W, IVS12nt1, R158Q, Y414C, IVS10nt546 гена ФАГ у 58 зразках з використанням специфічної ампліфікації 5, 7, 12 екзонів та 10, 12 інтронів та рестрикційного аналізу, двадцять три з яких у подальшому використовувались в якості ДНК-матриці для ампліфікації сьомого екзона гена ФАГ. 2. Підібрані умови для ампліфікації in vitro та рестрикції послідовності сьомого екзона гена ФАГ. 3. Розрахована математична модель відпалення праймерів та запропоновано оптимальний температурний режим ампліфікації, який можна представити у вигляді функції T=57.88+3.11e-0.0825τ. 4. Проведено аналіз алельного поліморфізму VNTR-локусу гена ФАГ з використанням специфічної ампліфікації in vitro послідовності 3'-нетрансльованої ділянки цього гену. 5. Проведені дослідження дозволили оптимізувати аналіз мутацій, що ідентифікуються для проведення пренатальної ДНК-діагностики фенілкетонурії з метою попередження народження хворих дітей.
![]()


![]() CAA, аргінін
CAA, аргінін![]() глутамін), R261X (CGA
глутамін), R261X (CGA![]() TGA, аргінін
TGA, аргінін![]() стіп кодон) і R261P (CGA
стіп кодон) і R261P (CGA![]() ССА, аргінін
ССА, аргінін![]() пролин). Детекцію перших двох представлялося можливим провести з використанням рестрикційного аналізу в ході даної роботи. Маркером мутації R261Q служить сайт рестрикції для ендонуклеази Hinf, що є присутнім у «нормальній» послідовності 7-го екзону гена ФАГ і відсутнім при мутації R261Q [74]. Аналогічна ситуація з рестриктазою Dde спостерігається при мутації R261Х [76].
пролин). Детекцію перших двох представлялося можливим провести з використанням рестрикційного аналізу в ході даної роботи. Маркером мутації R261Q служить сайт рестрикції для ендонуклеази Hinf, що є присутнім у «нормальній» послідовності 7-го екзону гена ФАГ і відсутнім при мутації R261Q [74]. Аналогічна ситуація з рестриктазою Dde спостерігається при мутації R261Х [76].
![]() ДНК.Пр (2.1)
ДНК.Пр (2.1)![]() (2.2)
(2.2) ![]() (2.3).
(2.3).![]() ДНК+Пр (2.4)
ДНК+Пр (2.4)![]() , (2.5)
, (2.5)![]() ДНК*.Пр (2.6)
ДНК*.Пр (2.6)![]() , (2.7)
, (2.7)![]() (2.8).
(2.8).![]() ДНК+Пр (2.9)
ДНК+Пр (2.9)![]() , (2.10)
, (2.10)![]() +
+![]() = З(ДНК)0 (2.11)
= З(ДНК)0 (2.11) 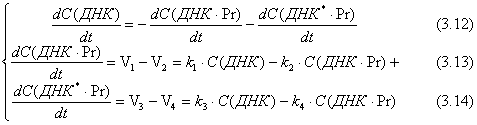
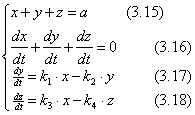
![]() (2.19)
(2.19) ![]() (2.20)
(2.20)![]() (2.21)
(2.21)![]() з рівняння (2.17) і одержимо рівняння (2.22) і (2.23).
з рівняння (2.17) і одержимо рівняння (2.22) і (2.23). ![]() (2.22)
(2.22)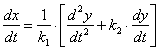 (2.23)
(2.23)![]() з рівняння (2.23) у рівняння (2.21) одержимо рівняння (2.24)
з рівняння (2.23) у рівняння (2.21) одержимо рівняння (2.24)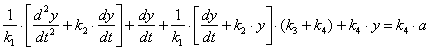 (2.24)
(2.24)![]()
![]() (2.25)
(2.25) ![]()
![]() (2.26)
(2.26)![]() (2.27)
(2.27)![]() .
.![]() (2.28)
(2.28)![]() (2.29)
(2.29)![]() (2.30)
(2.30)![]() , одержимо вираження (2.31).
, одержимо вираження (2.31).![]() (2.31)
(2.31)![]() (2.32)
(2.32)![]() (2.33)
(2.33)![]() (2.34)
(2.34)![]() (2.35)
(2.35)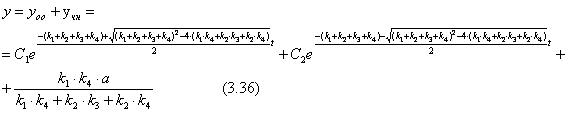
![]() з вираження (2.36).
з вираження (2.36).![]() (2.37)
(2.37)![]() у вираження (2.17) і виразимо x:
у вираження (2.17) і виразимо x: 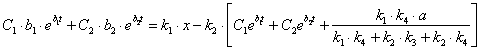 (2.38)
(2.38)![]()
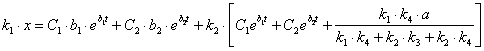 (2.38)
(2.38)![]() (2.39)
(2.39)![]()
 (2.40)
(2.40)
![]() , після підстановки у вираження (2.36) одержимо рівняння (2.37).
, після підстановки у вираження (2.36) одержимо рівняння (2.37).![]() (2.42)
(2.42)![]() , після підстановки у рівняння (2.40) одержимо:
, після підстановки у рівняння (2.40) одержимо: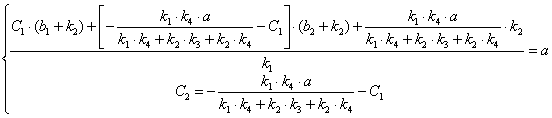 (2.43)
(2.43)![]()
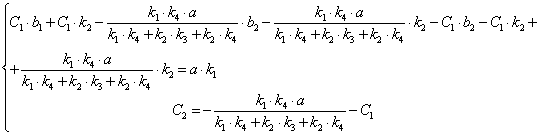 (2.44)
(2.44)![]()
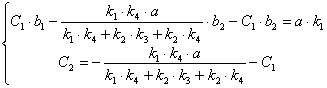 (2.45)
(2.45)![]()
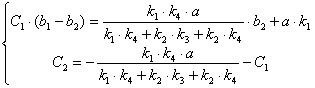 (2.46)
(2.46)![]()
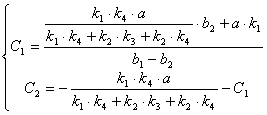 (2.47)
(2.47)![]()
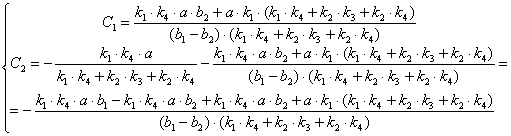 (2.48)
(2.48)![]()
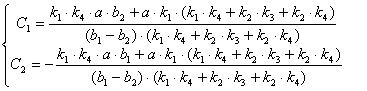 (2.49)
(2.49) (2.50)
(2.50)
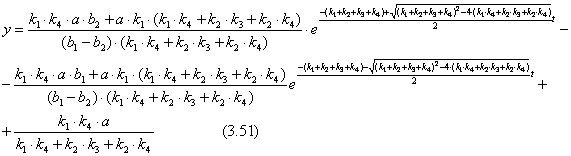

![]() ) може бути обчислена за допомогою константи рівноваги (K) з вираження (2.53).
) може бути обчислена за допомогою константи рівноваги (K) з вираження (2.53).![]() , (2.53)
, (2.53)![]() , (2.55)
, (2.55)![]() – зміна енергії Гібса в результаті реакції.
– зміна енергії Гібса в результаті реакції.![]() , (2.56)
, (2.56)![]() H – зміна ентальпії до і після реакції,
H – зміна ентальпії до і після реакції, ![]() S - зміна ентропії до і після реакції.
S - зміна ентропії до і після реакції.![]() (2.57)
(2.57)![]() (2.58)
(2.58)![]() з виражень (2.31-2.35):
з виражень (2.31-2.35):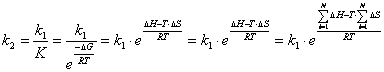 (2.59)
(2.59)![]() .
.![]() ) може бути розрахована за допомогою
) може бути розрахована за допомогою ![]() по тому ж алгоритму, що і
по тому ж алгоритму, що і ![]() , яка виражалась з
, яка виражалась з ![]() .
.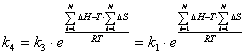 (2.60)
(2.60)![]() ,
, ![]() ,
, ![]() ,
, ![]() , a,
, a, ![]() та виразили
та виразили ![]() ,
, ![]() ,
, ![]() через
через ![]() , ми можемо побудувати графік зміни концентрації одноланцюгової ДНК (x = f1(
, ми можемо побудувати графік зміни концентрації одноланцюгової ДНК (x = f1(![]() )), комплексу ДНК-праймер (y = f2(
)), комплексу ДНК-праймер (y = f2(![]() )), неспецифічні комплекси ДНК-праймер z = f3(
)), неспецифічні комплекси ДНК-праймер z = f3(![]() ) з часом. Ми брали значення ентальпії і ентрапії з роботи [78]
) з часом. Ми брали значення ентальпії і ентрапії з роботи [78] ![]() ,
, ![]() ,
, ![]() . Даний графік представлений на рис. 2.12, розрахунки проводилися при температурі 60ºС. Для побудови графіка була використана програма Mathcard 2001 Прofessional.
. Даний графік представлений на рис. 2.12, розрахунки проводилися при температурі 60ºС. Для побудови графіка була використана програма Mathcard 2001 Прofessional.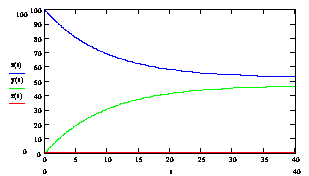
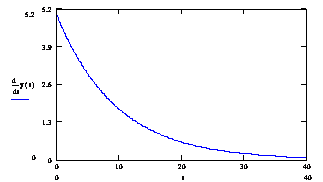
![]() ) у часі при температурі 60ºС.
) у часі при температурі 60ºС.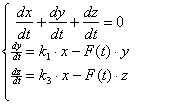 (2.61)
(2.61)![]() , чи
, чи ![]() , де
, де ![]() - температура,
- температура, ![]() - час. Однак рішення даного рівняння буде надзвичайно громіздким. Тому набагато простіше задатися температурою, що східчасто змінюється і при переході на нову температуру переносить попередні значення концентрації ssДНК, комплексів ДНК.Пр і ДНК.Пр*. Для цього ми можемо скористатися формулами (2.36, 2.40, 2.41) і задатися новими крайовими умовами y(0) = y0 і x(0) = x0.
- час. Однак рішення даного рівняння буде надзвичайно громіздким. Тому набагато простіше задатися температурою, що східчасто змінюється і при переході на нову температуру переносить попередні значення концентрації ssДНК, комплексів ДНК.Пр і ДНК.Пр*. Для цього ми можемо скористатися формулами (2.36, 2.40, 2.41) і задатися новими крайовими умовами y(0) = y0 і x(0) = x0. ![]() (2.62)
(2.62)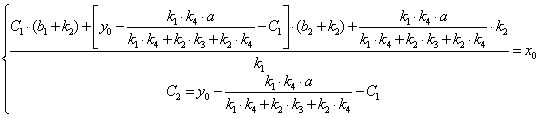 (2.63)
(2.63)![]()
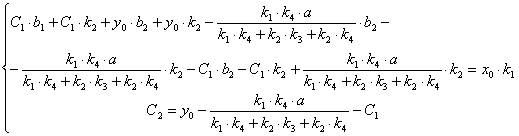 (2.64)
(2.64)![]()
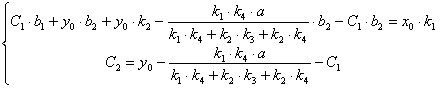 (2.65)
(2.65)![]()
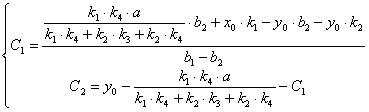 (2.66)
(2.66)![]()
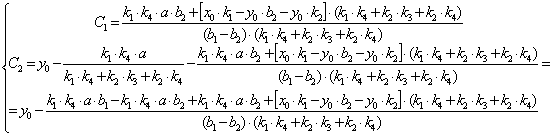 (2.67)
(2.67)![]()
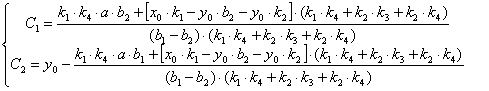 (2.68)
(2.68) (2.69)
(2.69)

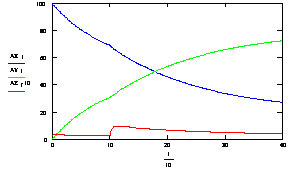
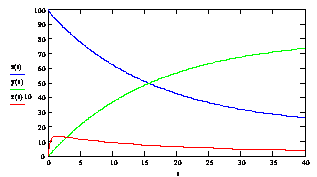
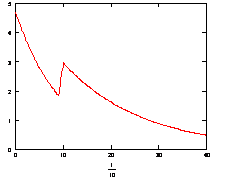
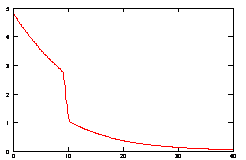
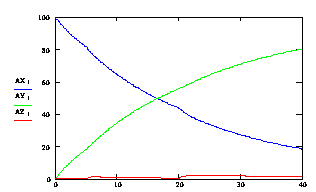
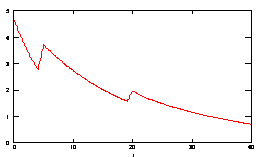
![]() ) у часі при температурі, що змінюється: 10 сек при 60°С, 30 сек при 58°С.
) у часі при температурі, що змінюється: 10 сек при 60°С, 30 сек при 58°С.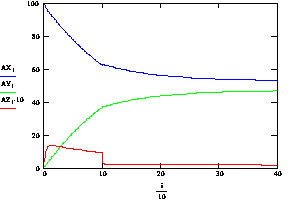
![]() ssДНК.Пр (2.72)
ssДНК.Пр (2.72)![]() ssДНК+Пр (2.73)
ssДНК+Пр (2.73)![]() ssДНК*.Пр (2.74)
ssДНК*.Пр (2.74)![]() ssДНК+Пр (2.75)
ssДНК+Пр (2.75)![]() ДНК + H3PO4 (2.76)
ДНК + H3PO4 (2.76)![]() ДНК* + H3PO4 (2.77)
ДНК* + H3PO4 (2.77)![]() ДНК (2.78)
ДНК (2.78)![]() (2.81)
(2.81) (2.82)
(2.82)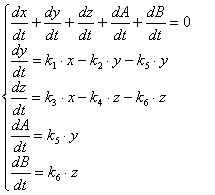 (2.83)
(2.83)![]() , а потім перераховуємо значення x, y, z, a, b при
, а потім перераховуємо значення x, y, z, a, b при ![]() c відповідно до системи рівнянь (2.84).
c відповідно до системи рівнянь (2.84).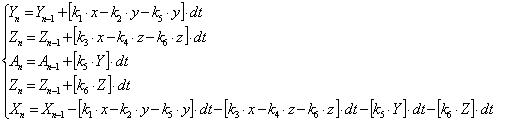 (2.84)
(2.84)![]() цих кроків ми одержуємо значення концентрацій, що відповідають 40с відпалу праймерів. Розрахунки проводяться за допомогою програми GraphBuilder, що була розроблена нами в середовищі програмування Borland Delphi 7.0. Даний програмний продукт інтегрований з базою даних Genome на базі MS SQL Server за допомогою технології Active Data Objects, у якій зберігаються дані про стан системи через кожні 100 млс, що еквівалентно десяти тисячам обчислень. Ці дані використовуються для побудови графіків.
цих кроків ми одержуємо значення концентрацій, що відповідають 40с відпалу праймерів. Розрахунки проводяться за допомогою програми GraphBuilder, що була розроблена нами в середовищі програмування Borland Delphi 7.0. Даний програмний продукт інтегрований з базою даних Genome на базі MS SQL Server за допомогою технології Active Data Objects, у якій зберігаються дані про стан системи через кожні 100 млс, що еквівалентно десяти тисячам обчислень. Ці дані використовуються для побудови графіків.
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]() (2.85)
(2.85)![]() (2.86)
(2.86)![]() (2.87)
(2.87)![]() (2.88)
(2.88)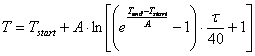 (2.89)
(2.89)
![]() (2.90)
(2.90)
![]() (2.91)
(2.91)